29.05.2024
Mittelinfrarot-Spektroskopie zur Echtzeitüberwachung eines Ultra-/Diafiltrationsprozesses
Dr. Simon Kern, Dr. Alexander Echtermeyer , S-PACT
Biopharmazeutika und Biotherapeutika gewinnen aufgrund ihrer spezifischen Anwendungen in der medizinischen Therapie zunehmend an Bedeutung. Diese Substanzen werden in der Regel durch die Kultivierung von Säugetierzellen hergestellt. Ein entscheidender Aspekt des Produktionsprozesses ist die Abtrennung und Reinigung dieser hochwirksamen Proteine.
Eine gängige Methode der Aufreinigung besteht darin, das Zielprotein zunächst anhand seiner Molekülgröße durch Ultrafiltration abzutrennen. Danach wird mittels Diafiltration - hier werden Salzgehalt oder Lösemittel der biomolekülhaltigen Lösung verändert - das Medium entsprechend den Vorgaben modifiziert. Abschließend folgt ein zweiter Ultrafiltrationsschritt, um die Zielkonzentration zu erreichen.

- Abb.1: Monipa®-System von IRUBIS
Überwachung biologischer Prozesse
Bei biotechnologischen Prozessen wird in der Regel Wasser als Medium im Überschuss verwendet, was zu vergleichsweise niedrigen Analytkonzentrationen führt. Insbesondere gilt dies für hochwirksame Proteine in Zellkulturen, die oft nur im niedrigen ppm-Bereich vorliegen. Direkte Analysen müssen daher hochspezifisch sein oder erfordern Vorkonzentrationsschritte.
Während die Raman-Spektroskopie den Vorteil der Unempfindlichkeit gegenüber Wasserinterferenzen bietet, was sie auch für stärker verdünnte Systeme anwendbar macht, wird die Mittelinfrarot-Spektroskopie weniger durch Farbe und Fluoreszenz beeinflusst, die typischerweise in biologischen Prozessen beobachtet werden. Bei Inline-Anwendungen lassen sich Messungen im mittleren Infrarot-Bereich am besten im Modus der abgeschwächten Totalreflexion (ATR) durchführen, da hier optische Weglängen von nur wenigen Mikrometern erreicht werden.
Messungen
Die Messungen werden in einer Ultrafiltrations-Diafiltrationsanlage (UFDF) durchgeführt, bei der der Retentat-Auslass des Membranmoduls direkt mit der Durchflusszelle in der Zuleitung verbunden ist (Abbildung 2).
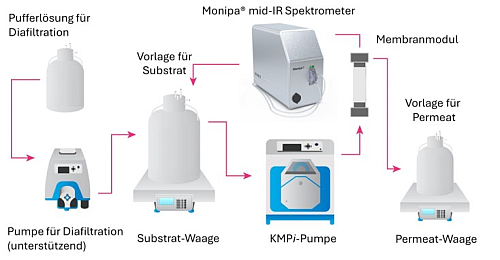
Abb.2: Schematische Darstellung eines UFDF-Aufbaus für die nachgeschaltete Proteinaufarbeitung [1].
(Mit Genehmigung von Alvotech Island.)
Jedes Spektrum wird über 60 Sekunden aufgezeichnet, um eine angemessene Dynamik für die Prozessüberwachung und -steuerung zu erhalten. Eine 3D-Spektrenübersicht eines vollständigen Filtrationsprozesses zeigt bereits die drei Stufen der Proteinkonzentration im Amid I+II-Bereich zwischen 1700 cm-1 und 1500 cm-1 (Abbildung 4).
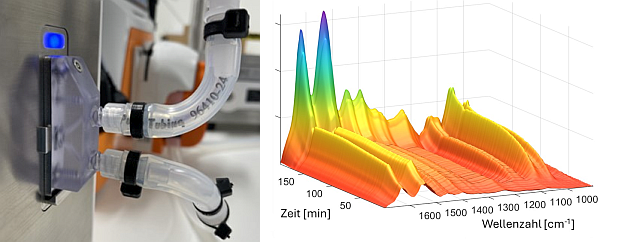
Abb. 3: 3D-gedruckte ATR-Durchflusszelle, montiert am Analysegerät. (Mit Genehmigung von IRUBIS und Alvotech.)
Abb. 4: 3D-Darstellung des spektralen Verlaufs eines dreistufigen Filtrationsprozesses.
Modellierung und Analyse
Die aufgenommenen Spektren sind von hervorragender Qualität und ermöglichen eine univariate - d.h. von nur einer Variablen abhängigen - Quantifizierung nach dem Lambert-Beer'schen Gesetz. Über die im Monipa®-Analysator eingebauten Kalibrierungen hinaus, die auf die Quantifizierung typischer Nährstoffe und Metabolite in biologischen Prozessen ausgelegt sind, können externe Software-Tools wie PEAXACT gängige chemometrische Ansätze für kundenspezifische Anwendungen ausnutzen. Im hier gezeigten Beispiel wird die numerisch integrierte Peakfläche durch einen Regressionsschritt mit einigen Proben bekannter Zusammensetzung in die Analyt-Konzentration umgerechnet (Abbildung 5).
Die Anwendung dieser Vorhersagemethode auf die Spektren des Monipa®-Analysators ermöglicht es, den Verlauf der Proteinkonzentration während des gesamten UFDF-Prozesses zu verfolgen. Die Messungen weisen ein bemerkenswertes Maß an Wiederholbarkeit auf, wodurch die Stabilität der vorhergesagten Profile über die Zeit gewährleistet ist. Diese Stabilität ermöglicht ein rechtzeitiges Eingreifen in den Prozess und/oder die Freigabe der Produktformulierung in Echtzeit (Abbildung 6)
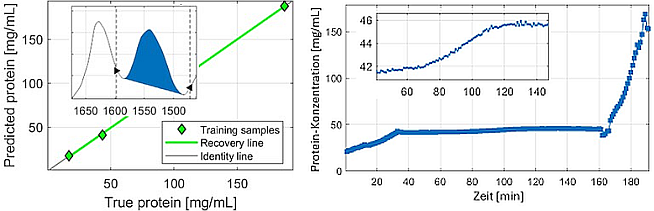
Abb.5: Paritätsdiagramm für eine 3-Punkt-Kalibrierung basierend auf der Integration der Amid-I-Bande bei 1540 cm-1 (Einschub).
Abb.6: Konzentrationsprofil eines vollständigen dreistufigen UFDF-Durchlaufs. Einschub: Detaillierte Ansicht der Diafiltrationsphase mit einer Wiederholbarkeit von nahezu ±0,1 mg/mL.
Zusammenfassung
Nicht-invasive spektroskopische Durchflussanalysatoren bergen ein erhebliches Potenzial für eine effiziente PAT-Überwachung und -Kontrolle sowohl in der vor- als auch in der nachgeschalteten biotechnologischen Verarbeitung (Upstream, Downstream). Der im gezeigten Beispiel verwendete, auf der Mittelinfrarot-Spektroskopie basierende, Analysator ist zur Überwachung der Ultrafiltrations-Diafiltrations-Aufarbeitung eines monoklonalen Antikörpers welcher als Biosimilar verwendet wird sehr gut geeignet. Die maßgeschneiderte ATR-Durchflusszelle liefert eine sehr gute spektrale Qualität und Stabilität, die eine unkomplizierte Anwendung von einfachen univariaten Quantifizierungsansätzen ermöglicht. Durch den Einsatz dieser Analysatoren in Produktionsumgebungen wird eine Produktfreigabe in Echtzeit möglich, wodurch das volle Potenzial der modernen PAT kommerziell genutzt werden kann.
Quellen
- Milewska A et al.; In-line monitoring of protein concentration with MIR spectroscopy during UFDF; (2023) Engineering in Life Sciences 23(2).
